DETAIL研究詳細
02海底堆積物中のDNA量で魚の個体数変動が捉えられることを初めて検証
- 環境DNA技術により海底堆積物から過去300年にわたる魚のDNAを発見!
- 魚の個体数変動が堆積物中のDNA量で捉えられることを世界で初めて検証
- 堆積物DNAによる地球上のあらゆる大型生物の長期動態の解明に期待
発表者
- 愛媛大学沿岸環境科学研究センター 准教授 加 三千宣(くわえ みちのぶ)
- 兵庫県立大学シミュレーション学研究科 准教授 土居秀幸
- 神戸大学大学院人間発達環境学研究科 准教授 源 利文
- 産業技術総合研究所地質総合研究センター 研究員 鈴木克明
発表のポイント
- 地球上の脊椎動物など大型生物のほとんどの種は、個体群の長期動態が不明である。
- 本研究では、過去の生物の動態解明に今後期待が寄せられる堆積物中の環境DNAに着目し、生物の個体数変動を推定する方法としての堆積物DNA技術の有用性について検討した。
- 別府湾から得られた堆積物中の魚のDNA量変化は、漁獲量変化と良い対応関係を示し、魚の個体数の長期動態が堆積物中のDNA量で捉えられることを世界で初めて検証した。
- 堆積物DNAを用いたモニタリング手法は、21世紀の地球上の大型生物のモニタリングを支える有用な技術として期待される。
発表の内容
背 景
地球上の脊椎動物など大型生物の個体群の動態の解明は、種の進化や絶滅、気候変動や人為撹乱に対する応答に対する理解、生物種の保全策にとって不可欠である。これまで生物学的モニタリングによってその個体数について明らかにされてきた。しかし、そうしたモニタリングデータは長いものでも100年前まで遡れる程度で、モニタリング以前の過去数百年、数千年という時間スケールの記録はない。一方、そうした長い時間スケールの個体数変動を記録している堆積物記録は、そのほとんどは湖や海の魚記録で、海では過去1000年を超える個体数変動記録は7分類群のみである。また、これらの記録が得られる水域もごく限られる。すなわち、地球上のほとんどのマクロ生物の個体群の長期動態は、いまだ謎に包まれているのが現状である。
本研究では、過去の魚の動態解明に今後期待が寄せられる堆積物中の環境DNAに着目し、日本列島周辺の有用浮魚類を対象に堆積物中にDNAが存在するかどうか、魚の個体数変動を推定する方法としての堆積物DNA技術の有用性について検討した。
研究内容
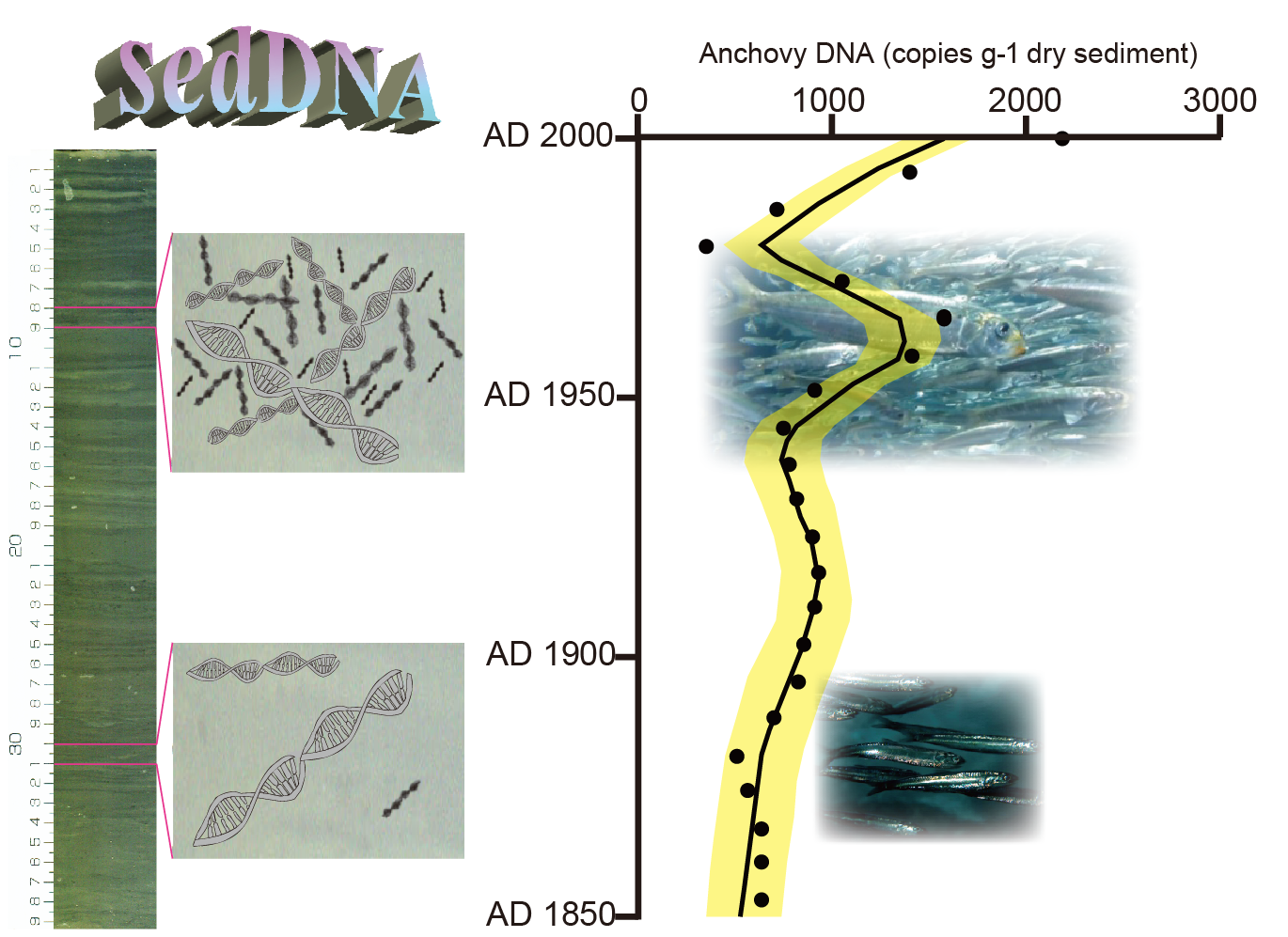
大分県別府湾で採取された約100cmの柱状海底堆積物試料を1cm間隔でスライスし、得られた湿潤試料を市販キットで精製した後、種特異的プライマーを使って定量PCR法によりDNAを定量した(図)。
その結果、堆積物からカタクチイワシ・マイワシ・マアジのDNAが検出され、300年前から現在までの海洋の堆積層中に魚のDNAが存在することが明らかとなった。また、そのDNA量の時系列変化は、数十年から数百年スケールで有意な変動を示し、漁獲量の記録との良い対応関係が認められた。さらに、過去300年間の魚鱗枚数との有意な関係も明らかになり、堆積物中の魚のDNA量が長期的な魚の個体数変動を反映していることがわかった。間隙水、魚鱗、魚骨、魚のペレット(糞)にDNAはほとんど含まれず、大部分は細粒サイズの粒子にDNAが吸着含有していることもわかった。
これらの結果は、堆積物のDNAを使って、魚の個体数の長期動態が復元できることを示唆している。
今後の展望
環境DNA技術は、近年比較的簡便・迅速且つ低コストの生物モニタリング法として有効性が認められ、世界各国の生物モニタリング手法としてスタンダードとなりつつある。しかし、こうしたモニタリングも始まったばかりで、そのデータ蓄積は10年にも満たない。しかも、そうした画期的なモニタリング技術も多くの研究者がいる欧米で国内の広域をカバーできても、地球上のすべてを網羅するには限界がある。一方で、堆積物DNAは、安定的に泥が堆積する水域があれば、その堆積物柱状試料から、100年分、1000年分の生物モニタリング情報が即座に得られることが期待される。しかし、堆積物中のDNA量が生物量や個体数を反映しているかについては明らかになっていなかったために、これまでの堆積物DNA技術は大型生物のモニタリングに利用されることはなかった。
本研究が示した結果は、個体群の生物量・個体数の長期動態の情報が長期保存されている堆積物柱状試料が得られれば、環境DNAによるモニタリングが始まる以前の生物情報だけでなく、これまでモニタリングでカバーできなかった地域の生物モニタリング情報の取得を可能にすることを示唆している。今後、地球上のマクロ生物の動態把握に、堆積物DNAが21世紀のモニタリングを支える有用な技術として期待される。さらに、過去の気候変動や人為撹乱による環境変動に対する生物の応答を詳しく調べることで、より確かな生物種の動態予測、生態系変化予測につながることが期待される。
発表論文
Michinobu Kuwae, Hiromichi Tamai1, Hideyuki Doi, Masayuki K. Sakata, Toshifumi Minamoto, and Yoshiaki Suzuki (2020) Sedimentary DNA tracks decadal-centennial changes in fish abundance. Communications Biology, doi: 10.1038/s42003-020-01282-9, (2020) October 8.
掲載学会誌/「コミュニケーションズ・バイオロジー」
研究成果名/ 堆積物DNAは魚種個体数の十年~百年スケール変化を捉えている
研究代表者/ 愛媛大学沿岸環境科学研究センター 准教授 加 三千宣
掲 載 日/ 令和2年10月8日
本研究の主な研究費
- 日本学術振興会科学研究費補助金 挑戦的萌芽研究 「堆積物中の環境DNAを用いた浮魚類の個体数復元に関する研究」
研究代表者 加 三千宣 (課題番号:18H01292) - 日本学術振興会科学研究費補助金 基盤研究 (B)(一般)
「沿岸堆積物コアでの環境DNAメタバーコーディングによる近過去魚類群集の復元」 研究代表者 土居秀幸
(課題番号:22340155) - クレスト(課題番号:JPMJCR13A2)
- 公益財団法人アサヒグループ学術振興財団環境研究助成 「浮魚類の環境DNAによる個体数復元に関する古海洋学的研究」
研究代表者加 三千宣